发酵乳制品已成为世界范围内人类日常饮食的重要组成部分,是现代生活中营养物质和有益微生物的良好来源。天然发酵乳制品是由原料乳微生物群与环境中存在的本土微生物共同作用而产生的。为加速发酵,最终采用接种技术,即“将少量前一阶段发酵液作为下一阶段发酵的原发酵剂”。该工艺自然选择了具有良好益生菌和生产性能的适应良好的微生物,能够缩短发酵时间,提高发酵品质。这种简单、可复制、可培养且易于操作的微生物生态系统是研究微生物群落形成机制和微生物遗传群体动态的绝佳环境,为了解复杂的微生物群内相互作用提供价值。
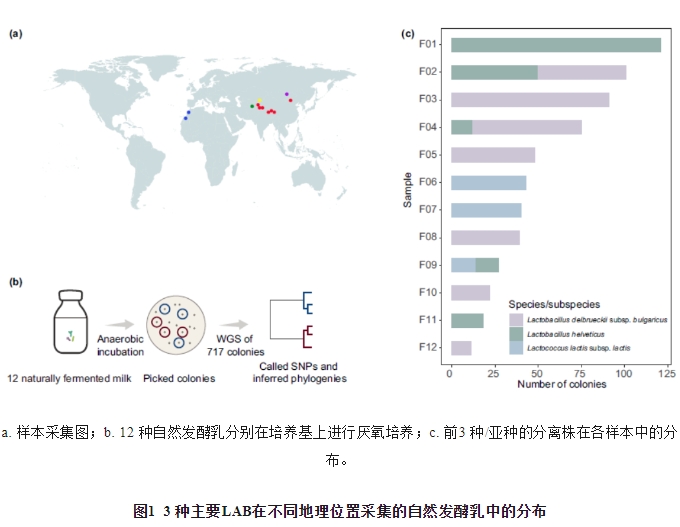
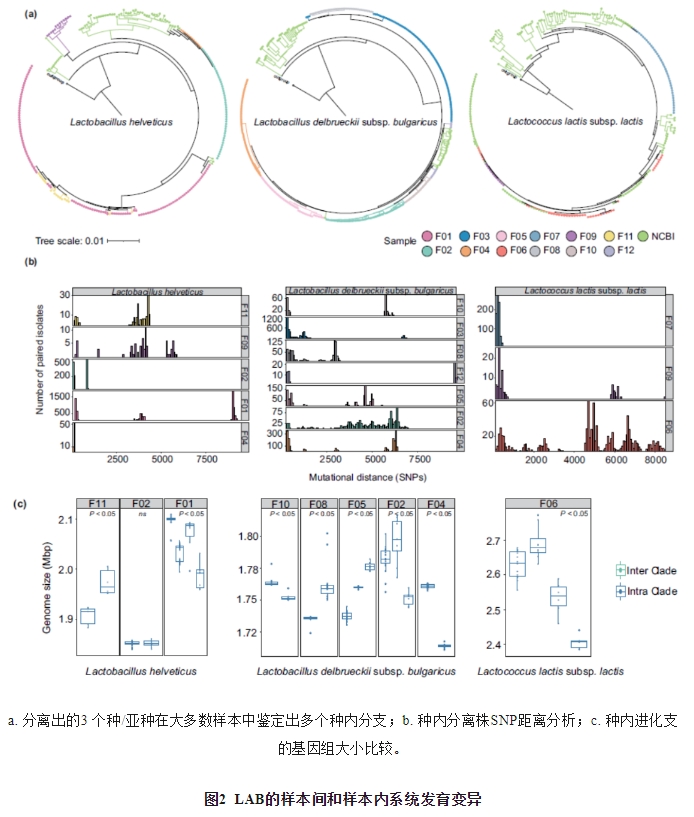
高通量测序技术的进步彻底改变了微生物种群的研究,包括发酵乳制品中的微生物种群。发酵乳制品中分离细菌的群体遗传学研究和宏基因组测序的使用,为发酵乳制品微生物的组成和功能潜力提供重要的见解。事实上,自然界中的微生物很少生活在孤立的生态位中;相反,它们与其他个体相互作用,形成复杂的群体。人们普遍认为,微生物相互作用对于维持微生物群落的稳定至关重要;然而,大多数相关研究都是在复杂的环境中进行的,并且仅限于物种水平的相互作用分析。在相对简单的实验系统(如传统的发酵乳)和菌株水平的遗传进化中,缺乏对物种间多样化的研究。较高的种内变异可能是由获得性突变和横向基因流动造成的,而种内多样性是由遗传漂变和来自生物和非生物环境的选择压力驱动的。有限的宏基因组测序深度和培养菌株的小样本量,一直是阻碍种内深度遗传进化分析的主要因素。宏基因组测序技术的突破大大降低了测序成本,提高了高通量测序的准确性。内蒙古农业大学乳品生物技术与工程重点实验室、农业农村部奶制品加工重点实验室、乳品生物技术与工程教育部重点实验室的You Lijun(第一作者)和张和平教授、刘文俊研究员(共同通信作者)旨在应用高质量的全基因组测序和配对宏基因组学分析研究菌株水平微生物群落形成的机制,并探讨微生物群内相互作用在驱动种内适应进化中的作用。研究分析从5 个国家(中国、吉尔吉斯斯坦、蒙古、摩洛哥和塔吉克斯坦)的12 个地区采集的12 种天然发酵乳,以确保采样的地理位置广泛,每个自然发酵乳样本都可以被视为一个独立的生态系统。生成了这些样品的深层宏基因组,样品包含相对简单的乳酸菌(lactic acid bacteria,LAB)群落。细菌培养3 个主要优势种/亚种,即瑞士乳杆菌(Lactobacillus helveticus)、乳酸乳球菌乳亚种(Lactococcus lactis subsp. lactis)和德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii subsp. bulgaricus)。因此,进一步分析了这3 个类群的样本内菌株水平的遗传进化。结果表明,LAB种群结构中有趣的样本间和样本内差异,其特征是高度的种内系统发育和功能基因组异质性,水平基因转移(horizontal gene transfer,HGT)是LAB基因获得的潜在机制。为了解天然发酵乳制品中LAB的生物地理多样性,从5 个亚洲和北非国家的12 个地区收集了12 个样本(图1a)。为对其进行分离,将等分的样品铺在厌氧环境下培养。随机选择具有LAB形态的单个细菌菌落进行分离,通过16S rRNA测序和全基因组测序进行鉴定(图1b)。最终,从7 个样品中分离培养出325 个L. delbrueckii subsp. bulgaricus,从5 个样品中分离培养出214 个L. helveticus,从3 个样品中分离培养出97 个L. lactis subsp. lactis(n=636)。探讨L. helveticus、L. delbrueckii subsp. bulgaricus和 L. lactis subsp. lactis样本内分离株的遗传进化关系。基于636 个分离株的核心基因和240 个公开保存的高质量基因组,重建自然发酵乳样品中每个种/亚种的分子系统发育(完整性>98%,污染<5%)。3 个种/亚种均观察到清晰的聚类模型。有趣的是,在大多数情况下,来自同一样本的多个同源株表现为单一谱系(例如,从F02、F04分离的L. helveticus;从F01、F02、F04分离的L. delbrueckii subsp. bulgaricus;从F03、F09分离的L. lactis subsp. lactis)这表明样品内的异位位点与来自不相关样品的异位位点相比,具有更密切的系统发育关系(图2a)。尽管样品内系统发育接近,但样品内每对分离物之间的变异距离仍然是可变的(L. helveticus、L. delbrueckii subsp.bulgaricus和 L. lactis subsp. lactis的单核苷酸多态性(single nucleotide polymorphism,SNPs)分别是4382、2418和2618)(图2b)。在大多数样本中,在每个种/亚种的样本内基因组大小中有显著差异(图2c)
基于最大相似法对样本中的3 个种/亚种的SNPs构建系统发育树(图3a,图5),并分析样品内进化支特异性肽酶和碳水化合物活性酶(carbohydrate-active enzymes,CAZyme)分布。样本F01中121 个L. helveticus基因组的SNP聚类分析,将分离物分为5 个主要的(A~E)和2 个次要的(F~G)系统发育支(图3a)。样品F01中121株L. helveticus的肽酶和CAZyme的平均数量分别为115和40(图3b)。主要支系的肽酶和酶的数量无显著性差异。基于L. helveticus 5个主要分支的肽酶和CAZyme分布,观察到显著的分支特异性聚类模式(肽酶:R2=0.56,P=0.001;CAZymes:R2=0.49,P=0.001,图3c、d),在进化支A和B中尤为明显,表明这2 种类型的基因在进化支间具有相当大的变异性。基因丰度分析鉴定出样品F01中5 个主要进化支的18 个差异肽酶和18 个CAZyme。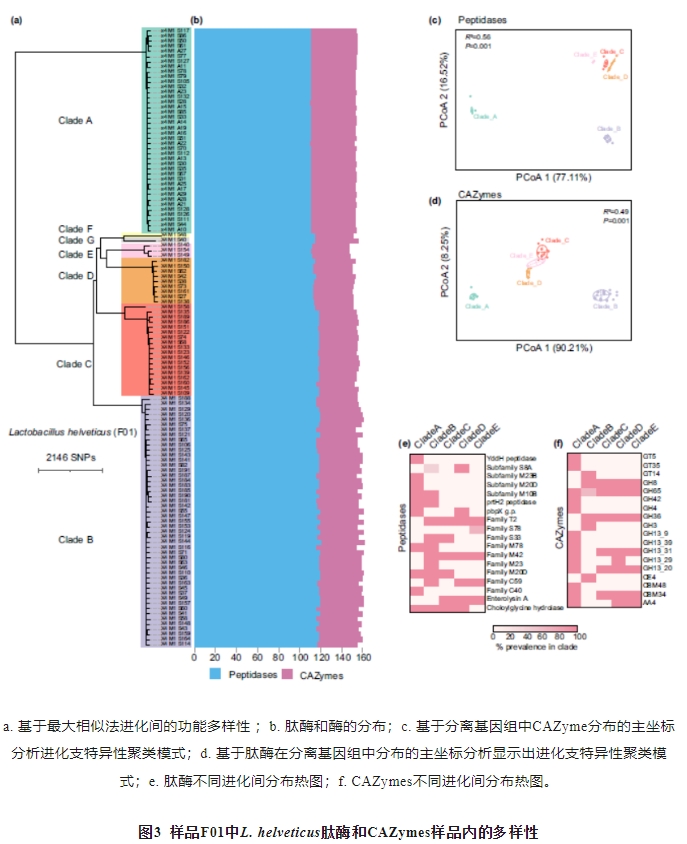
将进化支特异性基因的序列与相应的微生物发酵乳宏基因组(超500 个核苷酸具有99%识别度)进行比对,结果显示匹配率为36.36%~63.49%(包括样品F01分离出L. helveticus的407/641基因;F02分离出L. delbrueckii subsp. bulgaricus的64/176基因;F06分离出 L. lactis subsp. lactis的300/551基因。通过将这些匹配基因与非冗余(non-redundant,NR)蛋白数据库进行交叉比较(图4a)注释这些匹配基因,将它们分类为不可移动元件(non-mobile genetic elements,non-MGE)(包括编码假定功能或假设蛋白质的基因,84.38%~87.33%,F01分离出L. helveticus的351/407基因;F02分离出L. delbrueckii subsp. bulgaricus的54/56基因和F06分离出L. lactis subsp. lactis的262/300基因)和MGEs(包括转座子、质粒、噬菌体相关蛋白和其他可移动元件;12.67%~15.63%;F01分离出 L. helveticu的56/407基因;F02分离出L. delbrueckii subsp. bulgaricus的10/64基因;样品F06分理出L. lactis subsp. lactis的38/300基因)。牛乳宏基因组匹配的no-MGE进化支特异性基因的起源存在明显的种间差异(图4b)。这些基因中有很高比例被定位到同一物种的宏基因组序列中(从F01分离的L. helveticus的267/351基因,占76.07%;从F02分离的L. delbrueckii subsp. bulgaricus的25/54个基因,占比46.30%;从F06分离的L. lactis subsp. lactis2 的40/262基因,占91.60%。结果表明,它们可能是内源基因,存在于种/亚种内。其余与牛乳元基因组匹配的no-MGE支系特异性基因可能是由HGTs从其他密切相关的物种获得的了解LAB分离物中与LAB元基因组匹配的no-MGE进化源特异性基因的功能多样性,可以揭示驱动自然发酵LAB生态系统中高度特异性LAB群体适应性进化的选择性力量,通过NR蛋白和KEGG数据库对这些基因进行注释(图4c)。NR蛋白数据库注释(图4c和在线表S5)将这些基因中的大部分定位为假定的功能基因菌株(从F01中分离的L. helveticus的228/351基因,占比64.96%);F02中分离的L. delbrueckii subsp. bulgaricus的32/54基因,占比59.26%)。从F06分离的L. lactis subsp. lactis2 的176/262基因,占比67.18%。3 个种/亚种的KEGG注释基因呈现差异分布(图4d)。从F01中分离的L. helveticus在磷酸和氨基酸运输系统(11/351基因,3.13%)、碳固定(9/351基因,2.56%)和中央碳水化合物代谢(9/351基因,2.56%)中富集。F06中分离的L. lactis subsp. lactis富含糖、多元醇和脂质转运系统(10/262基因,占3.82%)和ABC-2型等转运系统(8/262基因,占3.05%)。F02中分离的L. delbrueckii subsp. bulgaricus只含有16 个功能上属于KEGG亚模块的基因,并且没有发现特别富集的KEGG模块。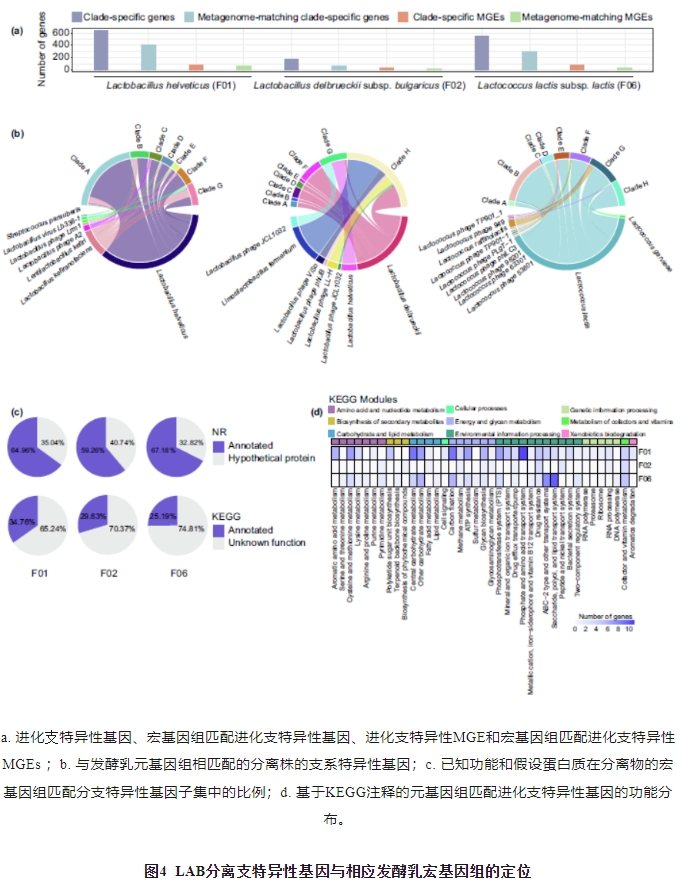
共鉴定出327 种,其中主要种13 种(平均相对丰度>1%,图5a),占总种数的87.92%;以L. delbrueckii (25.59%)、L. helveticus(16.68%)、Streptococcus thermophilus(15.55%)和L. lactis(9.58%)为主。12 种天然发酵乳的发酵乳微生物群均以LAB为主,但其物种水平的微生物群组成差异较大(图5a),这可能与原料、发酵剂接种量、生产环境、生产方法、地理位置等因素有关。比较L. helveticus、L. lactis subsp. lactis和L. delbrueckii subsp. bulgaricus中检测到的SNPs及宏基因组对应体相异,分离基因组中检测到的SNPs少于宏基因组,分别占SNPs平均比例的60.99%、85.57%和51.18%;但2 组SNP数据呈显著正相关(r=0.59,P=0.02,图5 b)。虽然2 个数据集检测到的SNPs的绝对数量存在差异,但显著的相关趋势表明这3 个种/亚种微生物宏基因组的SNPs得到了合理的表征。基因组π是衡量群体内多态性程度的群体遗传指标,以每个样本中所有可能的个体对之间SNP数的平均成对差异计算。3 个种/亚种的宏基因组相对丰度与π值呈较强的负相关(r=-0.65,P<0.008),这表明随这3 个种/亚种在发酵乳微生物群中所占比例的增加,它们的基因组变异性降低,种群稳定性增加(图5c)。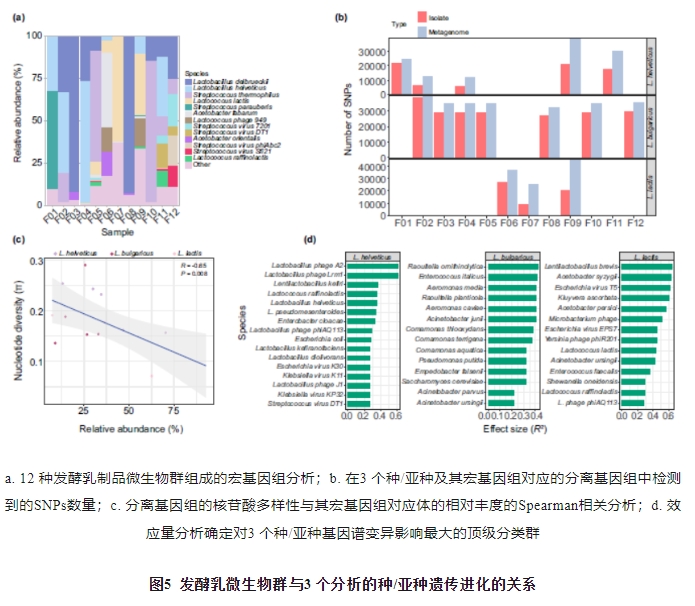
研究自然发酵乳制品中具有代表性的3 种菌种,即Lactobacillus helveticus、Lactococcus lactis subsp. lactis和Lactobacillus delbrueckii subsp. bulgaricus。通过结合LAB分离株的全基因组测序和发酵乳样品的深度宏基因组测序,分析揭示了这3 个种/亚种在样品内复杂的种内遗传和功能多样性。此外,样品的微生物组成和潜在的样品内水平基因转移是影响菌株多样性的重要因素。总之,生态和遗传因素共同塑造了自然发酵乳制品生态系统中的种内微生物异质性。
文章《Intraspecific microdiversity and ecological drivers of lactic acid bacteria in naturally fermented milk ecosystem》发表于Science Bulletin 2023年58卷内蒙古农业大学张和平教授、刘文俊研究员为本文共同通信作者文章链接:https://doi.org/10.1016/j.fbio.2023.103159
免责声明:本文仅代表作者个人观点,与中国益生菌网无关。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。
版权声明
1.本站部分转载的文章非原创,其版权和文责属于原作者。2.本网所有转载文章、链接及图片系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与我们联系,对可以提供充分证据的侵权信息,bio149将在确认后12小时内删除。3.欢迎用户投递原创文章至86371366@qq.com,经审核后发布到首页,其版权和文责属于投递者。