Nature子刊 | 利用肠道宏基因组测序数据或可量化宿主信息?
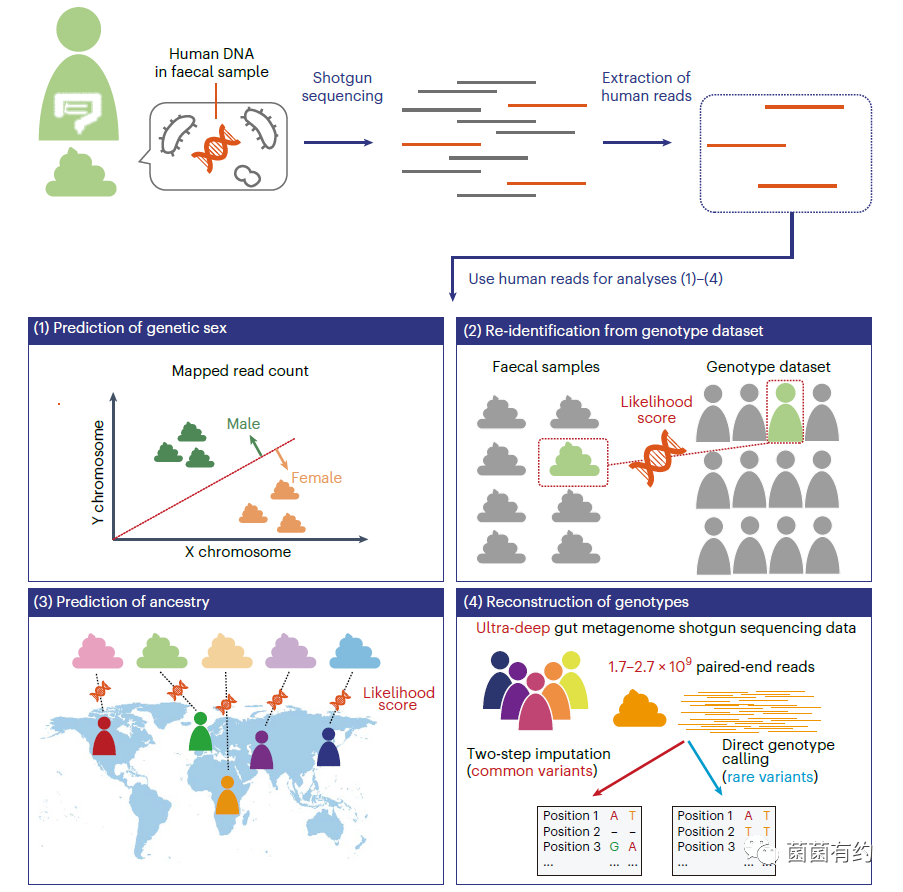
时间:2023-05-25 来源:菌菌有约 作者:小菌君 浏览次数:1599随着测序技术和生物信息学的快速发展,极大地促进了研究人员对人体微生物组的解析。已知粪便样本中存在的人类DNA会导致肠道宏基因组测序数据中混杂少量人的测序reads(约10%左右),经常为减少分析干扰及保护捐赠者的隐私而被去除,在宏基因组分析流程中常简称“去宿主”。然而,目前尚不清楚从这种reads中可重建多少个人信息,且这还没有得到定量评估。通过定量评估不仅有利于澄清与数据共享有关的伦理问题,还能借助粪便样本获取人类基因信息用于研究和取证。近日,日本大阪大学研究人员在Nature Microbiology发表最新研究,从343名日本人粪便宏基因组中重建了个人信息,并获得了相关的人类基因型数据。
内容要点·
1:从343名日本人肠道宏基因组中重建个人信息,其中97.3%的样本可根据性染色体的测序深度准确预测遗传性别;
2:基于从粪便宏基因组数据中恢复的人类reads,借助可能性评分能从匹配的基因型数据中重新识别个体,灵敏度为93.3%,且该方法也能够预测98.3%的个体的祖先;
3:对5个粪便样本进行超深层宏基因组测序,并对血液样本进行全基因组测序,使用基因型calling法,证明常见和罕见变体的基因型都可从粪便样本中重建(包括临床相关的变异);
4:尽管存在伦理问题,但本研究中使用的重新鉴定方法有助于对肠道宏基因组和人类种系基因型数据集进行质量控制。
小菌君读后感
肠道宏基因组数据中的人类reads对于粪便取证、检出稳健变异和估计多基因疾病风险意义重大(例如 2 型糖尿病); 小菌君团队最近发现对于低生物量样本(如阴道样本),宏基因组测序的宿主率高达80-90%,如何进一步利用高宿主率样本,解析宿主和菌群间的关系仍需深入探索和思考!
免责声明:本文仅代表作者个人观点,与中国益生菌网无关。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。
版权声明
1.本站部分转载的文章非原创,其版权和文责属于原作者。2.本网所有转载文章、链接及图片系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与我们联系,对可以提供充分证据的侵权信息,bio149将在确认后12小时内删除。3.欢迎用户投递原创文章至86371366@qq.com,经审核后发布到首页,其版权和文责属于投递者。







