导读
基于宏基因组现有资源已经揭示了人类肠道微生物组的多样性和功能,但由于基因组质量不足以及缺乏来自典型研究不足人群的样本,对其进一步的了解受到限制。本研究使用混合长读长PromethION和短读长HiSeq测序来表征60名内蒙古人(三个时间点,180个样本)的粪便微生物群,这些样本是乳双歧杆菌Probio-M8酸奶干预实验的一部分。本研究构建了内蒙古肠道基因组(IMGG)数据库,包括802个环状和5927个高质量的宏基因组组装基因组(MAG)。该方法提高了基因组的组装连续性及基因组元件的分辨率,包括核糖体RNA操纵子(rrns)、代谢基因簇(MGCs)、前噬菌体和插入序列(IS)。特别是,本研究报道了未培养物种的rrn拷贝数、超过12000个先前未发现的肠道前噬菌体以及IS在肠道细菌中的分布。总体而言,该数据为进一步研究人类肠道微生物群提供了高质量、大规模的资源。
论文ID
原名:A high-quality genome compendium of the human gut microbiome of Inner Mongolians
译名:内蒙古人肠道菌群高质量基因组集合
期刊:Nature Microbiology
IF:30.964
发表时间:2023.1.5
通讯作者:张和平 & 孙志宏
通讯作者单位:内蒙古农业大学
DOI号:10.1038/s41564-022-01270-1
实验设计
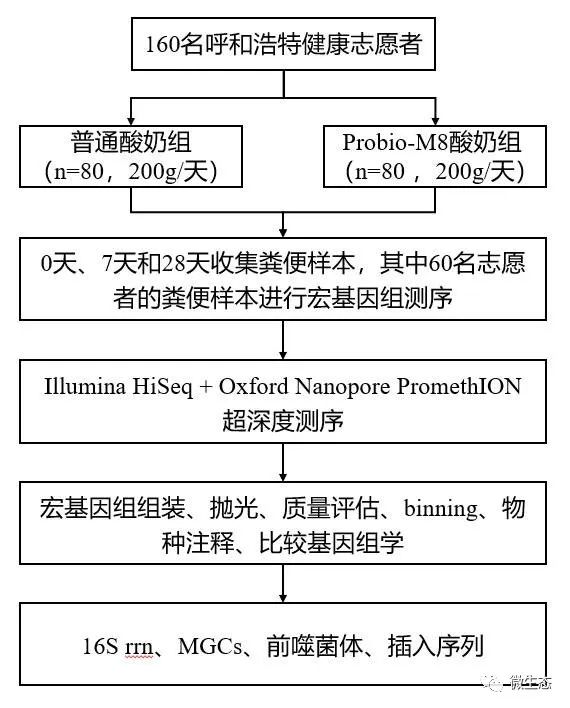
结果
本研究使用了先前进行的一项基于生物标记物的纵向人群实验,该实验研究了与普通酸奶相比,每天摄入益生菌酸奶(含有乳双歧杆菌Probio-M8,Probio-M8)的有益效果。180份粪便样本(60名受试者;三个时间点:开始食用酸奶后0天、7天和28天)在PromethION和HiSeq平台上进行了超深度宏基因组测序,共生成3.7 Tbps三代和20.1 Tbps二代数据(即每个样本20.5±4.5 Gbps和111.8±8.65 Gbps;补充表1)。三代数据的质量远远超过了大多数现有的人类肠道三代数据库(补充表2),平均N50长度为8千碱基对(Kbps);平均reads长度为6 Kbps;平均reads质量为9.5。超深度混合基因组测序获得了平均N50长度为278 Kbps的基因组合集(116 Kbps~1420 Kbps,仅包括组装最小长度≥ 10 Kbps,即所有组装基因组的98.5%),对应的平均组装长度为314 Mbps(82~564 Mbps;补充表3)。最大contig为6.77 Mbps,6688个contig大于1 Mbps(每个样本:平均值±标准差=37.16±12.64 contig,8~74个contig)。为了评估组装流程的准确性,本研究基于三种测序和组装策略,直接从摄入益生菌酸奶的个体粪便样本中组装Probio-M8的基因组:即仅三代序列组装、二三代混合组装、仅二代序列组装。对于每个组装方法,重建50个Probio-M8基因组,并与Probio-M8参考基因组进行比较(补充表4)。如预期一样,与单独使用二代或三代测序构建的基因组相比,二三代混合组装的基因组在多个基因组质量参数方面表现出了极大的改进,包括组装连续性、碱基对和基因预测准确性、基因组完整性、全基因组相似性度量以及功能注释的精确度(补充图1)。此外,二三代混合组装的基因组组装错误率与二代数据覆盖率呈负相关,但与测序深度无关(补充图2)。因此,二三代混合组装是一种强有力的策略,可以直接从复杂的人类肠道宏基因组数据库组装环状和更加准确的基因组。本研究由混合测序组装的高质量基因组共产生了802个CMAG(补充表5)。从复杂的微生物群中组装CMAG对于大多数宏基因组研究来说都是一个巨大的挑战,在撰写本手稿时,仅有225个CMAG包括在公共基因组数据库中(补充表6)。其中大多数(n=160)是从环境宏基因组中还原的;其中一些属于候选门级辐射类群,其成员通常为一个约1 Mbp的小基因组。而本研究中组装的CMAG较大(1.0~5.5 Mbps(平均约2.4 Mbps));该范围涵盖了大多数已知肠道细菌的基因组大小。为了评估当前组装的CMAG代表性,以95%的ANI阈值进行物种水平基因组去冗余,然后将我们的数据库与人类胃肠基因组(UHGG)数据库中的基因组(去冗余)进行比较。共有134物种水平的CMAG,涵盖11个门、14个纲、27个目、45个科和94个属(图1a;补充表5)。值得注意的是,131个物种水平的CMAGs是其物种下首个环状、完整和人类特有的代表性基因组(截至2021年7月),其中65个未培养物种的参考基因组质量得到了提升。例如,在UHGG数据库中,仅有15个高度碎片化的基因组Victivallis sp002998355,在本研究组装了该物种的完整及具有代表性的基因组,该基因组具有高分辨率的多拷贝区域,包含核糖体RNA操纵子(rrns)、IS和前噬菌体等元件(图1b)。本研究中组装的另一物种水平的CMAG是Bacteroides_A plebeius_A(一个已培养物种,图1c)。尽管有数百个公共数据库含有该物种的基因组和17个分离株,但其可用序列的N50长度从未超过150 Kbps。这些结果表明了当前工作流程在从复杂的宏基因组数据库和极具组装挑战的区域中组装完整基因组方面的能力和有效性。
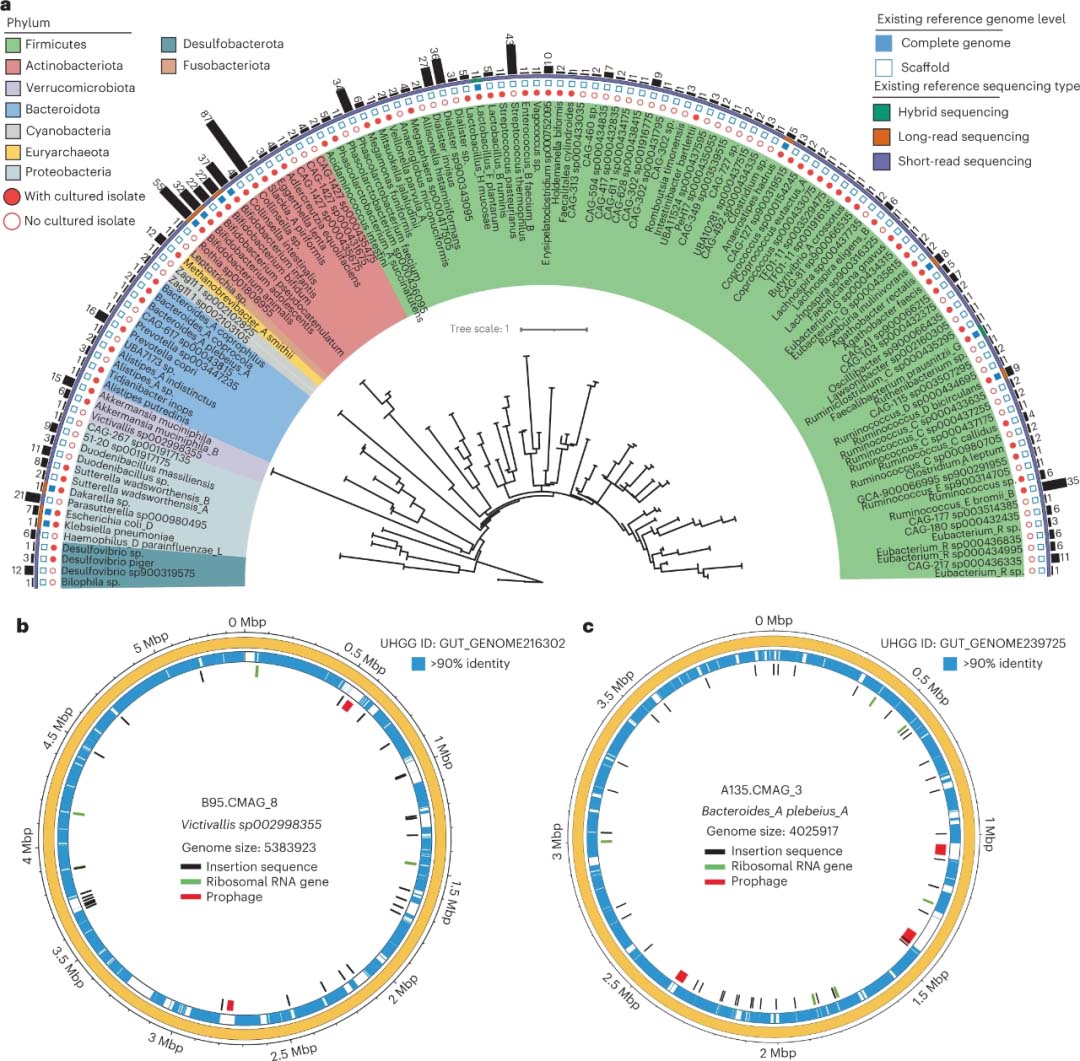
图1 有效组装大量物种水平的完整(环状,无间隙)宏基因组组装基因组。a,从本研究中组装的134个具有代表性的物种水平完整(环状,无间隙)宏基因组组装基因组(CMAGs)构建的系统发育树。门水平、已培养分离株的可用性、现有参考基因组的水平和测序类型由各自的颜色代码和符号表示。条形的高度加上外圆旁边的数字表示每个物种水平CMAG的组装基因组数量。b-c,两个CMAG的基因组示意图。最外面的圆(第1个):刻度(bps);第二圈:两个当前组装的CMAG,(b)B95.CMAG_8和(c)A135.CMAG_3;第三圈:最接近的基因组同源物,即Victivallis sp002998355和Bacteroides_A plebeius_A。>90%的区域以蓝色显示。单个scaffold中的插入序列、核糖体RNA基因和前噬菌体的位置分别用黑色、绿色和红色表示。
3 、内蒙古高质量肠道基因组数据库
初始bins由两种binning方法(即MetaBAT、vamb)生成,然后通过DASTool和内部脚本进行整合和优化。加上上述的CMAG,本研究总共还原了12391个MAG,符合MIMAG标准中的中等质量标准(>50%完整度和<5%污染率;补充表7)。事实上,其中6729个MAG符合MIMAG标准中列出的更严格的高质量定义(即,完整度>90%,污染率<5%,5S、16S和23S rRNA基因和至少18个tRNA基因;即IMGG数据库)。接着,对IMGG数据库中的6729个MAGs和从UHGG中检索到的147835个高质量基因组(完整度>90%,污染率<5%)进行聚类分析(图2a)。聚类过程产生了包含至少一个IMGG的485个宏基因组物种(MGS)。还原的MGS在分类上分为11个门、14个纲、30个目、40个科,涵盖220个属(图2b;补充表8)。大多数属于厚壁菌(72.7%),其次是拟杆菌(14.9%)、放线菌(4.8%)和变形菌(3.9%)。在485个MGS中,469个是UHGG和IMGG数据库共有的。有趣的是,这469个普通MGS来源于UHGG数据库中75.6%的高质量MAG,尽管完整的UHGG数据库包含3000多个不同物种,表明它们是人类肠道中的高丰度物种。其余16个MGS是IMGG数据库特有的,在分类上被分为五个不同的门,其中一半属于梭菌目(补充表9)。本研究的工作流程显著改善了MAG的连续性(图2c),IMGG数据库中的contig数量和N50长度均显著高于UHGG数据库(两种情况下均为P<2e-16)或代表性基因组(contig数量,P<1.6e-128;N50长度,P<3e-113)。此外,尽管UHGG数据库包含相当多的MAGs(n=111744),但只有5053个符合MIMAG标准中规定的高质量标准,UHGG数据库中的大多数分离株(n=4058)都是已培养的,这意味着基因组质量需要进一步改善。另一方面,这项研究为288种物种提供了具有代表性的高质量MIMAG参考基因组,154种物种中可用的高质量MIMAG的数量增加了50%以上,包括UHGG数据库中一些高度代表的物种,如Agathobacter rectalis、Alistipes putredinis、Bacteroides_B dorei和Lachnospira eligens_B(图2d;补充表8)。
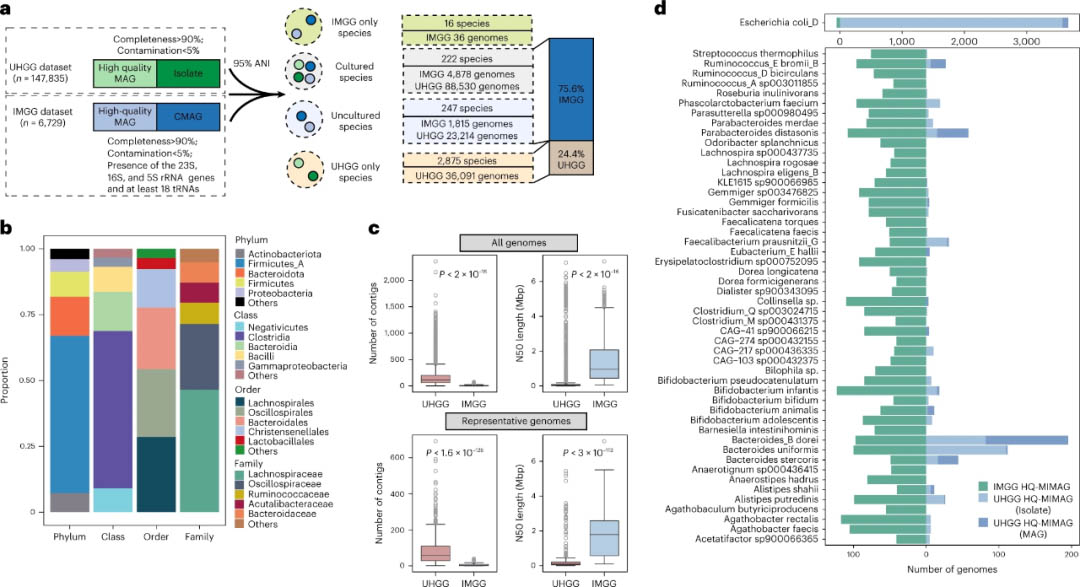
图2 内蒙古人肠道基因组IMGG数据库是一个扩展的基因组资源。a,6729个IMGGs(内蒙古人肠道基因组数据库)和147835个UHGGs(统一人类胃肠道基因组数据库)的聚类分析结果概述,阈值为95%的平均核苷酸一致性(ANI)。75.6%的UHGGs基因组与IMGGs具有显著同源性(>95% ANI),其余24.4%的UHGGs基因组与IMGGs数据库具有独特的同源性。b,IMGG数据库在门、纲、目和科水平的分类学分布。在每个分类水平中只显示前五个,其余的被定义为“其他”。c,UHGG(共计147835个基因组,485个代表基因组)和IMGG(共计6729个基因组,485个代表性基因组)数据库的基因组组装性能(contig数量和N50长度)比较。统计学差异用Wilcoxon秩和检验(双侧)。方框表示四分位范围,方框内的线表示中位数,须表示1.5倍四分位范围内的最低值和最高值。d,IMGG数据库中50个最大的物种簇现有的高质量MIMAG(宏基因组组装基因组的最小信息)数量。
4 、提高复杂肠道基因组区域的分辨率
为了揭示复杂的肠道基因组区域(包括rrns、MGC、前噬菌体和IS),将从IMGG数据库组装的485个MGS与UHGG数据库中的对应物进行了比较。首先,对rrn进行分析,因为它们是基于短读长组装的基因组中经常缺失的基因元件。特别是,16S rrn是未培养微生物生态学中不可缺少的分子标志物;然而,其可变的基因拷贝数常常导致微生物丰度分析中的严重偏差。正如预期那样,与UHGG相比,IMGG有着更多的rrn拷贝(包括16S、23S和5S rRNA;P<2e-16)(图3a);并且,IMGG和NCBI基因组数据库中对应的完整分离基因组的rrn拷贝数基本一致(扩展数据图1),这表明先前严重低估了UHGG数据库中MGS的rrn基因拷贝数。此外,通过对最全面的基因拷贝数数据库(rrnDB)进行分类搜索,其中85%(413/485)当前组装的MGS是找不到的,包括一些16S基因拷贝数高的未培养MGS(例如,Romboutsia timonensis、Intestinibacter bartlettii和Clostridium sp.;补充表10)。这些结果表明,由于基因组质量不足,目前可用的基因拷贝数参考数据库在很大程度上仍然不完整。其次,使用gutSMASH(一种专门用于分析肠道微生物群数据集的MGC预测工具)比较IMGG和UHGG中MGC的完整性。研究结果表明,在UHGG中发现的大部分MGC(56.5%)位于contig边缘,可能不完整,而IMGG中的比例很低,只有4.5%(图3b)。此外,在IMGG和UHGG数据库中分别发现了每个基因组具有6个和3个完整的MGC,这表明在IMGGs中对于识别全长的MGC有了实质性改善(图3b)。IMGG中包含MGC编码序列的基因组比例明显高于UHGG(P<7e-18;图3c),特别是在几个MGC类别中,包括非蛋白源氨基酸、芳香族和其它短链脂肪酸(SCFA)(图3d)。值得注意的是,MGC区域的基因组比例仅与短读长基因组组装的N50长度相关,而与IMGG无关(IMGG和UHGG的重复测量相关性分别为R=0.03和0.19;图3e)。表明MGC识别和分配的有效性和解决方案受到短读长数据中组装连续性不足的限制。第三,与UHGG数据库相比,IMGG数据库中前噬菌体基因组的分辨率有了很大提升。我们的方法在95%的IMGG中都能检测到前噬菌体序列(与UHGG(55%)相比)。IMGG和UHGG每个基因组编码前噬菌体的平均数量分别为4个和1个。IMGG中的前噬菌体序列的连续性也较高,N50长度为37974 bp(UHGG中为31064 bp;图3f);IMGG中前噬菌体编码区的基因组比例是UHGG的4.2倍(P<1e-77;图3g)。与UHGG相比,IMGG中的IS区域更加精确,反映在每个物种的IS区域总拷贝数(增加了三倍;P<1e-51;图3h)和IS编码区域的基因组比例(增加了近四倍;P<9e-57;图3i)。IS类别中改善程度最高的是IS1380(与UHGG相比,改善了5.6倍),其次是IS5、IS1634和ISAS1(分别是5.5倍、5.5倍和5.3倍;图3j)。
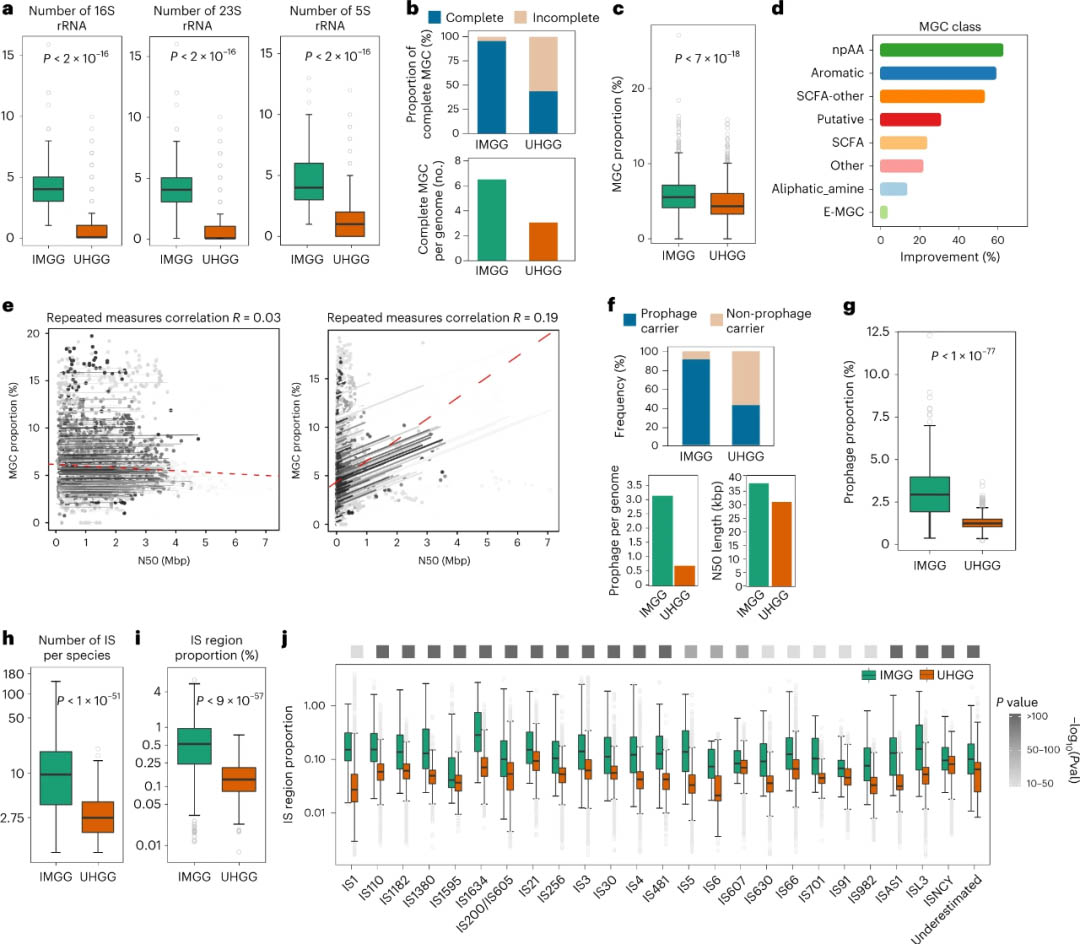
图3 内蒙古人肠道基因组增强遗传元件的基因组分辨率。内蒙古人肠道基因组(IMGGs;n=6729)和统一人类胃肠道基因组(UHGGs;N=147835)数据库。a,rRNA基因拷贝数。b-d,代谢基因簇(MGC)的恢复性能通过图b中MGC的完整水平和每个基因组中完整的MGC数量来评估;c,物种水平上MGC编码区在基因组中的比例(IMGG和UHGG数据库的数量分别为482和456);d,与UHGG相比,IMGGs各类MGC编码区比例的提高。MGC的预测由gutSMASH进行,根据产物将MGC分为不同的基因簇类:non-proteinogenic氨基酸(npAA);苯的硝基衍生物(Aromatic);SCFA与另一分子结合产生(SCFA-other);未知功能的基因簇(Putative);脂肪酸最多含有5个碳原子(SCFA);未分类的通路(Other);其中至少一个H被烷基取代基取代的氨衍生物(Aliphatic_amine);与能量捕获机制有关(E-MGC);e,IMGGs(左图)和UHGGs(右图)的MGC编码区比例与基因组组装N50长度之间的重复测量相关性(rmcorr)。灰色和红色虚线表示每个物种的相关性和所有物种的总体相关系数,通过R包(rmcorr)计算。IMGG和UHGG数据库中前噬菌体和插入序列IS的比较:f,前噬菌体载体的频率,每个基因组的前噬菌体数量和N50长度;g,在物种水平上,前噬菌体序列区域在基因组中的比例(IMGG和UHGG数据库分别为449和408);h,每个物种的IS数量;图i整体和图j单个IS在基因组中的区域比例(IMGG和UHGG数据库中n=485)。统计学差异用Wilcoxon秩和检验(双侧)。对于所有箱线图,框表示四分位区间,框内的线表示中位数,须表示1.5倍四分位区间内的最低值和最高值。
5 、人类肠道微生物中高度多样化和分化的MGC
深入研究可用高质量基因组中编码的代谢潜能。考虑到由于组装连续性有限,基于短读长的MGC分析的性能较低,本分析仅包括UHGG和IMGG数据库中的高连续性和高质量基因组(contig数<30)。共从15512个基因组中还原了97428个MGC区域,其中78675个是完整的,并被纳入进一步分析。大约一半的完整MGC(48%)与已知MGC没有同源性(70%的相似性阈值)。这些完整的MGC属于所有八个MGC类别,分布在58个MGC类型中(补充表11)。最常见的类别是SCFA(40.0%),其次是Putative(28.9%)和E-MGC(17.2%)(扩展数据图2)。然后,门水平分布和聚类分析揭示了MGC类别的总体分布在门水平的显著差异(图4a),以及在十个主要的门之间,它们在不同门中的数量各不同(图4a)。此外,基于涵盖九个主要门的功能性MGC分类分布的特异性聚类表明,MGC概述和组成在门水平间存在显著差异(Adonis检验R=0.38,P<0.001;扩展数据图3)。富集/缺失分析均显示,优势门中某些功能类别的丰度存在显著差异(图4b)。SFCA代谢相关的MGC在较低分类学水平的分布分析发现:两个优势目,Lachnospirales和Oscillospirales,包含大多数SCFA生物合成通路(图4c);每个未培养物种都有3~6个SCFA生物合成MGC。这些未培养物种中的一些最近发现了以前未报告的Christensenellales和Oscillospirales分类群,突出了这些代表性不足的分类群在促进宿主健康方面的潜力(图4c)。大约12%的MGC携带多个核心功能域(称为混合MGC)。混合MGC显著大于单功能域MGC(P<2e-16;扩展数据图4a)。最常见的混合MGC组合是琥珀酸-丙酸和红杆菌固氮(Rnf)复合物(扩展数据图4b)。最大的混合MGC大小为117 Kbps(B46.bin_5_Region10,由Flavoniflactor plautii物种携带;图4d)。它包含五个MGC功能域,其中两个属于E-MGC和SCFA其他类别(分别为Rnf复合物和乙醇胺利用[EUT]通路),占MGC总长度的80%以上;另外三个交错的功能域(焦磷酸硫胺素[氨基酸代谢]、未分类的脂肪酸和黄素酶脂质分解代谢)属于假定类。这些结果表明,肠道微生物组MGC编码广泛的代谢潜能,不同关键人类肠道分类群的代谢潜能差异很大。这项研究为系统揭示人类肠道代谢潜力提供了一个起点。
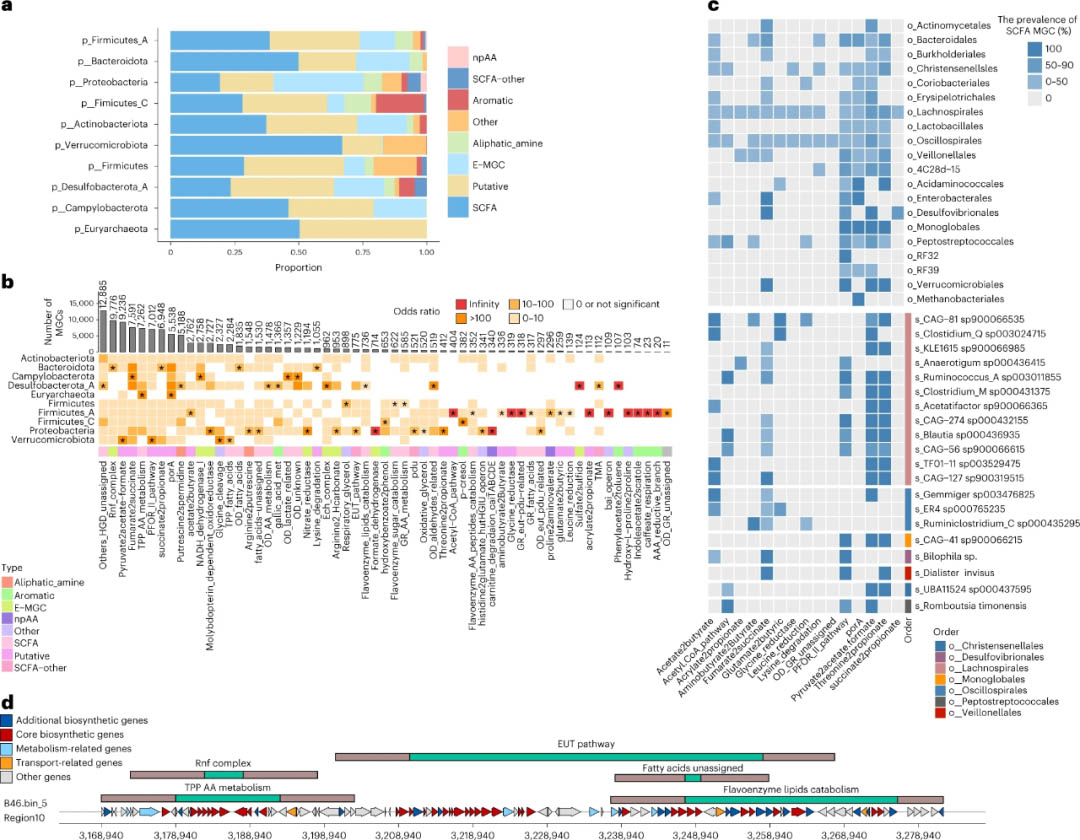
图4 人肠道菌群中代谢基因簇库的概述。a,不同种类的代谢基因簇在优势门水平的分布。MGC的预测由gutSMASH进行,根据产物将MGC分为不同的基因簇类:proteinogenic氨基酸(npAA);苯的硝基衍生物(Aromatic);SCFA与另一分子结合产生(SCFA-other);功能未知的基因簇(Putative);脂肪酸最多含有5个碳原子(SCFA);未分类的通路(Other);其中至少一个H被烷基取代基取代的氨衍生物(Aliphatic_amine);与能量捕获机制有关(E-MGC)。b,顶部图显示了完整MGC的数量。底部图为MGC型按门富集的热图;该门中某一特定MGC类型的显著富集用星号表示(P<0.05,Fisher检验)。比值表示特定MGC富集或短缺的程度,颜色强度越高表示富集越强,反之亦然。c,低分类学水平(优势目和未培养种)短链脂肪酸相关MGC的分布分析。d,gutSMASH预测的最大的混合型MGC(约117 Kbps)含有5个MGC功能域,可能编码不同的代谢通路。Rnf复合体,Rhodobacter固氮复合体;TPP AA代谢、硫胺素焦磷酸(氨基酸代谢);EUT通路,乙醇胺利用通路。
6 、12834个肠道前噬菌体的分类和功能注释
前噬菌体是另一类被低估的肠道微生物群。因此,本研究随后挖掘了IMGG数据库中未知的肠道前噬菌体。IMGG数据库中总共还原了21217个前噬菌体基因组,根据未培养病毒基因组(MIUViG)最低信息标准,将其进一步聚类为13437个物种水平的病毒操作分类学单位(vOTUs;补充表12)。这些vOTU代表39839 Kbps的N50长度。接下来,使用科水平和物种水平的系统发育距离,通过与宏基因组肠道病毒(MGV)数据库进行比较,对vOTU进行分类。值得注意的是,95.5%(n=12834)的vOTU与MGV数据库在物种水平不具有同源性。除了那些可分类到物种水平(4.5%)的病毒外,90.9%的vOTU与MGV水平具有科水平的同源性,但只有46.7%的病毒被分类到已知的病毒科(图5a)。大多数已分类的前噬菌体属于Siphoviridae和Myoviridate科(分别为73.0%和24.2%;图5b)。进一步分析表明,最常见的细菌宿主是Firmicutes_A(71.4%),这也是先前未报告前噬菌体的主要宿主。Siphoviridae和Myoviridate都是人类肠道中的优势前噬菌体科,其宿主范围广泛,涵盖多个门(图5c);然而,Siphoviridae(Firmicutes_A [68.8%],Bacteroidota [15.58%],Actinobacteriota [5.3%])和Myoviridae [Firmicuters_A[83.0%]的主要细菌宿主在门水平的分布却有很大差异。如预期那样,crAssphage的宿主范围小且具有特异性,仅由拟杆菌属组成。接下来,对IMGG数据库中的前噬菌体基因组进行深入分析,以探究其功能能力。从12834个物种水平的vOTU代表性基因组中鉴定出596193个蛋白质编码基因,并将这些假定基因与几个常见的功能和/或病毒注释数据库进行了比较。结果表明,仅有55.4%的前噬菌体基因在功能上能够被分类,而44.6%的基因与任何交叉比较的数据库都不具有同源性,被分配给了未知的功能(图5d)。而这些不匹配的基因代表了大量的病毒功能基因和代谢能力未被开发。大约8.88%的功能基因编码辅助代谢通路(排名前三的通路:氨基酸代谢,2.51%;辅因子和维生素代谢,1.81%;碳水化合物代谢,1.55%;图5e)。值得注意的是,辅助代谢通路在前噬菌体科之间的分布各不同(图5e)。CrAssphage具有较高比例的辅因子和维生素代谢基因,而微小病毒科具有较高水平的参与能量代谢和氨基酸代谢通路的基因(图5e)。多糖降解基因占所有酶的22%,它们主要属于未分类的前噬菌体、丝状病毒科和肌病毒科(图5f)。然后,预测IMGG数据库中的假定抗生素耐药基因(ARGs)。能检测到的ARGs占总预测基因的0.15%(n=916),远高于先前研究(0.0028%)。共有712个前噬菌体基因组携带ARGs;其中大多数与四环素、大环内酯和丁胺卡那霉素有关(图5g),分别占检测到的ARGs的43.1%、31.2%和9.7%。这些结果表明,尽管携带ARG的噬菌体很少存在于肠道微生物中,但这些噬菌体可能编码ARG(例如,A163.CMAG_1_1_7_59800编码假定的β-内酰胺酶基因;图5h),可能成为ARG转移的关键载体,并对人类健康造成严重威胁。这些结果表明,肠道前噬菌体在分类和功能上比先前我们所了解的更加多样化。IS是最常见的转座元件,它能够在宿主基因组内剪切和整合,在细菌适应和进化中发挥着难以置信的作用。然而,在基于短读长的MAG中,它们常被忽略。因此,我们从公共数据库(补充表13)和IMGG数据库中基于长读长的MAG中检索到的肠道细菌分离株的完整/染色体水平基因组,确定了IS元件的综合信息。共鉴定出140336个IS(属于26个IS家族)。最常见的IS是IS3(17.0%),其次是IS200/IS605(9.3%)和IS256(8.1%),而最不常见的是ISKRA4(仅0.02%;补充表14)。不同IS在多个门中分布不同(图5i)。例如,弯曲杆菌属和脱硫杆菌属的IS200/IS605比例都很高,但它在其他门中的普遍度较低。值得注意的是,在所有门中,变形菌门和弯曲杆菌门分别具有最多和最少的IS拷贝数(图5i)。此外,IS拷贝数的MAG内整体的高变异性(扩展数据图5)表明拷贝数变异在人类肠道微生物群中广泛存在。转座元件携带转座酶编码基因,也可能携带非转座酶基因,即乘客基因。为了探索可转座元件的功能潜能,对乘客基因(位于IS边界内)和邻近基因(位于靠近IS,在5000 bp内)进行了分析和注释。我们的结果表明,20.2%的IS编码至少一个假定的功能基因,乘客基因总数为36308个,平均每个IS携带0.26个。IS21拥有最多的功能性乘客基因(占总乘客基因的17.3%),其次是IS3和IS66(分别为17.0%和12.4%;图5j)。UniProt数据库注释显示,这些乘客基因在DNA整合、细胞活性、转录调节、DNA重组和DNA复制中发挥作用(图5k),尽管其中大多数(48.4%)被分配为“非特征化蛋白质”(补充表15)。使用KEGG数据库对IS区域附近的基因和通路进行注释发现,它们与广泛的功能相关,包括遗传信息处理、信号和细胞过程、代谢、碳水化合物代谢、信号转导、膜转运、细胞群落-原核生物、核苷酸代谢、翻译和氨基酸代谢(扩展数据图6)。
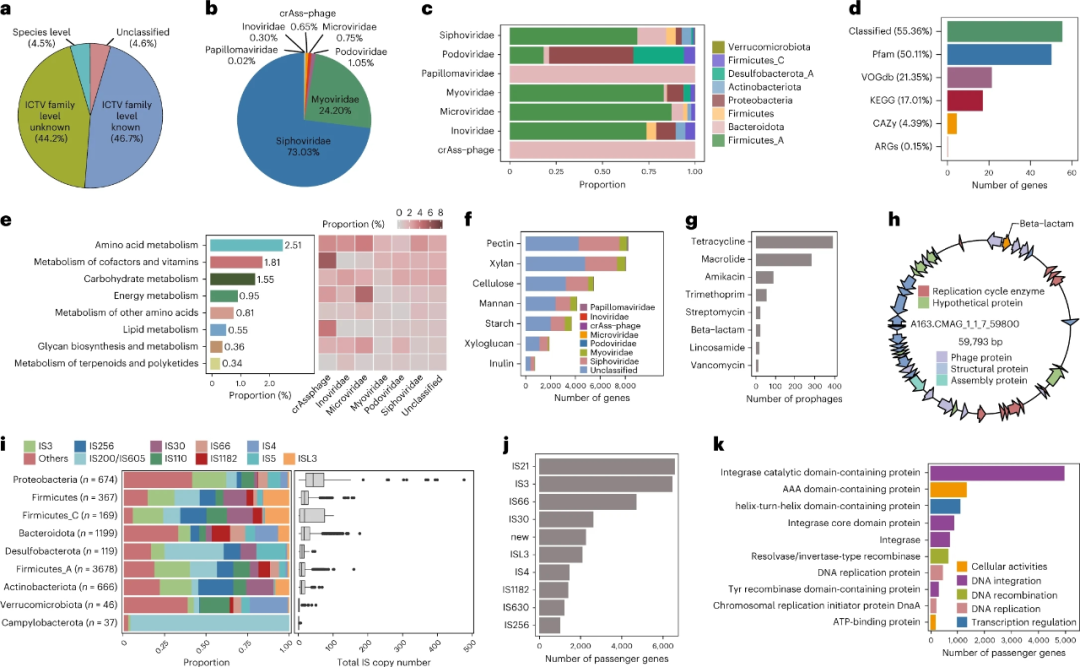
图5 肠道中前噬菌体和插入序列的概述。a,对13437个病毒操作分类单元(vOTU)与宏基因组肠道病毒(MGV)数据库进行同源性搜索,并根据国际病毒分类委员会(ICTV)的病毒分类概要进行分类。b,已知前噬菌体的科水平分类注释与分布。c,主要前噬菌体科在不同菌门中的分布。d,通过5个常见的功能和/或病毒基因数据库/软件注释的假定前噬菌体基因,包括蛋白质家族数据库(Pfam)、京都基因和基因组百科全书(KEGG)、病毒同源数据库(VOGdb)、碳水化合物活性酶(CAZY)和ABRicate(用于抗生素耐药基因的预测,ARGs)。搜索可发现55.36%的功能分类的前噬菌体基因,而剩下的44.64%是未知的。e,参与辅助代谢通路的基因比例及其在病毒科中的分布。f,不同底物的多糖降解基因在不同前噬菌体科水平的分布。g,ARGs数量。h,含有一个假定内酰胺酶基因的前噬菌体的例子。i,左图:插入序列(IS)在细菌门水平的分布。右图:每个门的总IS拷贝数。j,携带最多乘客基因的十大IS。k,通过UniProt数据库注释揭示了乘客基因最常见的生物学功能。
讨论
在这项研究中,我们使用混合长读长PromethION和短读长HiSeq测序对60名内蒙古个体(三个时间点,180个样本)的粪便微生物群进行了表征。我们构建了IMGG数据库,包括802个环状的和5927个高质量的基因组,这些基因组符合MIMAG标准中规定的高质量基因组标准,极大地扩展了人类肠道高质量MIMAG的现有数据库(n=7492)。此外,据我们所知,这是迄今为止最大的CMAG基因组数据库,是目前可用CMAG的三倍多。因此,IMGG数据库是一个有价值的基因组资源。基于该方法,实现了基因组连续性的提高,从而提升了复杂基因组区域中基因组元件的分辨率,包括rrns、MGC、原噬菌体和IS。尽管这些区域在细菌适应和进化中具有重要的功能,但由于缺乏高质量的参考基因组,尚未设计出针对性分析它们的系统和具体研究。IMGG数据库为在rrnDB中未发现的430种物种提供了相对准确的16S rRNA基因拷贝数信息,这表明目前可用的rRNA基因拷贝数数据库和基于16S rRNA的功能预测工具在很大程度上是不完整的,应该扩展到覆盖未描述和/或未培养的物种。然后,在IMGG数据库中将MGC功能分配给肠道细菌发现,提高基因组的连续性大大提高了预测的MGC的完整性,揭示了人类肠道微生物群中MGC的多样性和差异性比以前想象的更广泛。基因组连续性的增加也有助于MGC的鉴定,特别是具有多个功能域的混合MGC。尽管MGC可以容易地组装到仅具有短读长的MAG中,但基因组连续性不足严重限制了对特定未充分探索人类肠道分类群中MGC功能的解读,特别是对于靠近边界的MGC。IMGG数据库的病毒组分析显示,大量前噬菌体与目前已知的物种没有同源性/同源性低。这些肠道前噬菌体编码重要的功能基因,如辅因子和维生素代谢途径、多糖降解酶和ARGs,支持肠道病毒在人类健康和营养中的关键作用。事实上,前噬菌体被认为是肠道微生物之间基因转移的关键载体,这进一步表明了它们在肠道微生物群落中一些关键功能基因移动中的重要性。此外,对人类肠道病毒的彻底了解将是开发治疗方法(例如,基于噬菌体的微生物组策略)以对抗微生物失调相关的慢性和/或变性疾病的先决条件。本研究强调了人类肠道微生物群相关IS中存在广泛的物种间多样性和拷贝数变异,但却很难参透,因为现有基因组中重复区域的分辨率有限。本研究中揭示的IS特征进一步表明它们参与了基因组结构的重塑,并对微生物群的适应性产生影响。有趣的是,少数IS甚至含有假定的功能基因。这些潜在的乘客基因可能在微生物之间迅速而广泛地传播,这可能会在暴露于环境选择力时影响微生物基因组的稳定性。除了IS元件中携带的乘客基因外,许多假定的功能基因也在IS附近富集。先前的研究表明,IS可以插入功能基因并影响其活性。因此,IS通过调节其相邻基因的功能和表达来影响微生物活性的潜在生理作用值得进一步研究。长读长测序技术(即PacBio HiFi测序和新型牛津纳米孔测序技术)的持续进步可以减少容易出错的长读长并提高组装的精确度,有望避免或至少减少未来对深度互补短读长的需求。总的来说,我们的结果支持二三代混合组装是一种有效的策略,可以提高直接从复杂人类肠道宏基因组中组装的MAG的连续性、准确性和分辨率。使用当前的方法,本研究构建了IMGG数据库,这是一个高质量基因组的巨大集合,对代表性不足的基因组区域进行了细致观察。可以设想,该数据库和其他大规模的高质量基因组数据库可以在未来整合,为统一质量的基因组构建存储库,进一步实现基因组解析的宏基因组学。
原文链接:https://www.nature.com/articles/s41564-022-01270-1
免责声明:本文仅代表作者个人观点,与中国益生菌网无关。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。
版权声明
1.本站部分转载的文章非原创,其版权和文责属于原作者。2.本网所有转载文章、链接及图片系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与我们联系,对可以提供充分证据的侵权信息,bio149将在确认后12小时内删除。3.欢迎用户投递原创文章至86371366@qq.com,经审核后发布到首页,其版权和文责属于投递者。













