传统发酵乳制品的宏基因组特征
时间:2021-12-09 来源:益生之源 作者:尤利军 浏览次数:29812021年12月8日,内蒙古农业大学:尤利军(1作),杨成聪(共1),张和平*(通讯)等在LWT-Food Science and Technology(IF:4.952)发表题为“Metagenomic features of traditional fermented milk products”的研究文章。

结果表明,不同类型样品间的微生物群落组成差异较大,特别是优势物种。此外,基于宏基因组组装方法,73份样品中共得到153个基因组(MAGs),包括四个假定的新物种(分别隶属于乳杆菌属,链球菌属,醋酸杆菌属和罗氏菌属),它们的基因组中包含大量编码细菌素的基因,可能与发酵过程不良微生物的控制有关,同时还鉴定到碳水化合物活性酶和次级代谢物簇的各种基因元素,这些元素可能影响乳制品的风味,质量和/或安全性。此外,宏基因组分析解析了发酵乳中噬菌体的含量与细菌间的相互作用,尤其是噬菌体的侵染史。噬菌体可能在形成传统发酵乳微生物群组成中发挥了重要作用,例如Myoviridae的相对丰度低于25%时,其与Lactobacillaceae呈负相关,但其相对丰度超过25%时,则呈正相关。宏基因组深度测序能够发现更多的发酵乳环境微生物群落的宏基因组特征 (MAGs个数,CDS编码基因个数、质粒个数和水平基因转移频率)。
综上, 本研究强调了传统发酵乳中微生物多样性和异质性。我们的研究结果表明宏基因组深度测序对了解低复杂度微生物群落是十分必要的。
原文链接:
https://doi.org/10.1016/j.lwt.2021.112945
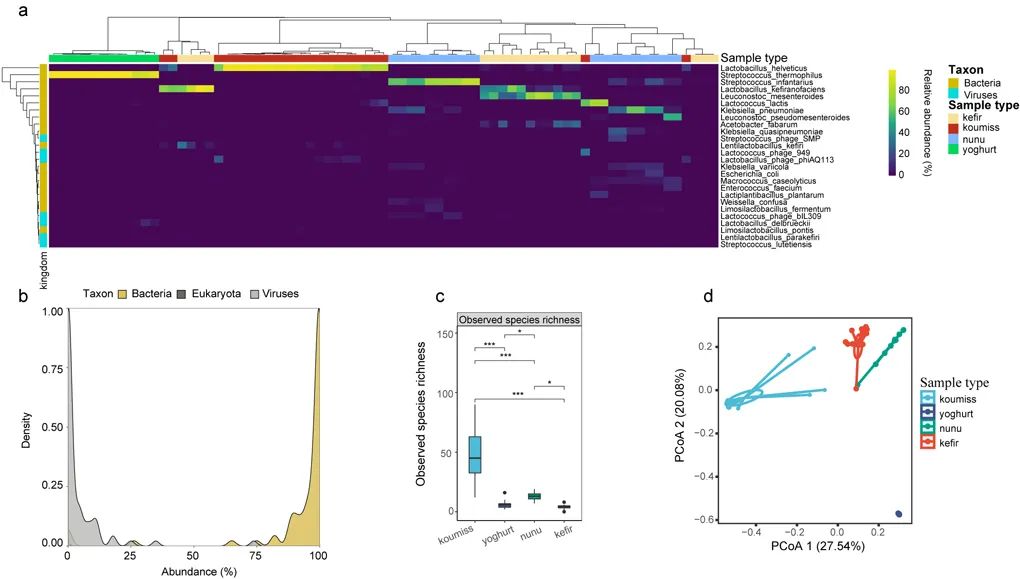
传统发酵乳制品群落组成及多样性
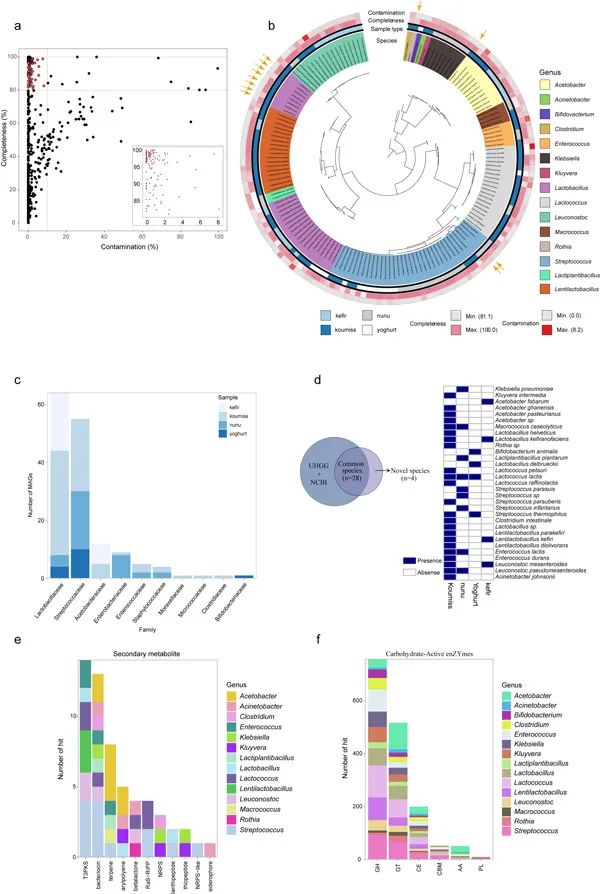
传统发酵乳制品宏基因组组装与分析
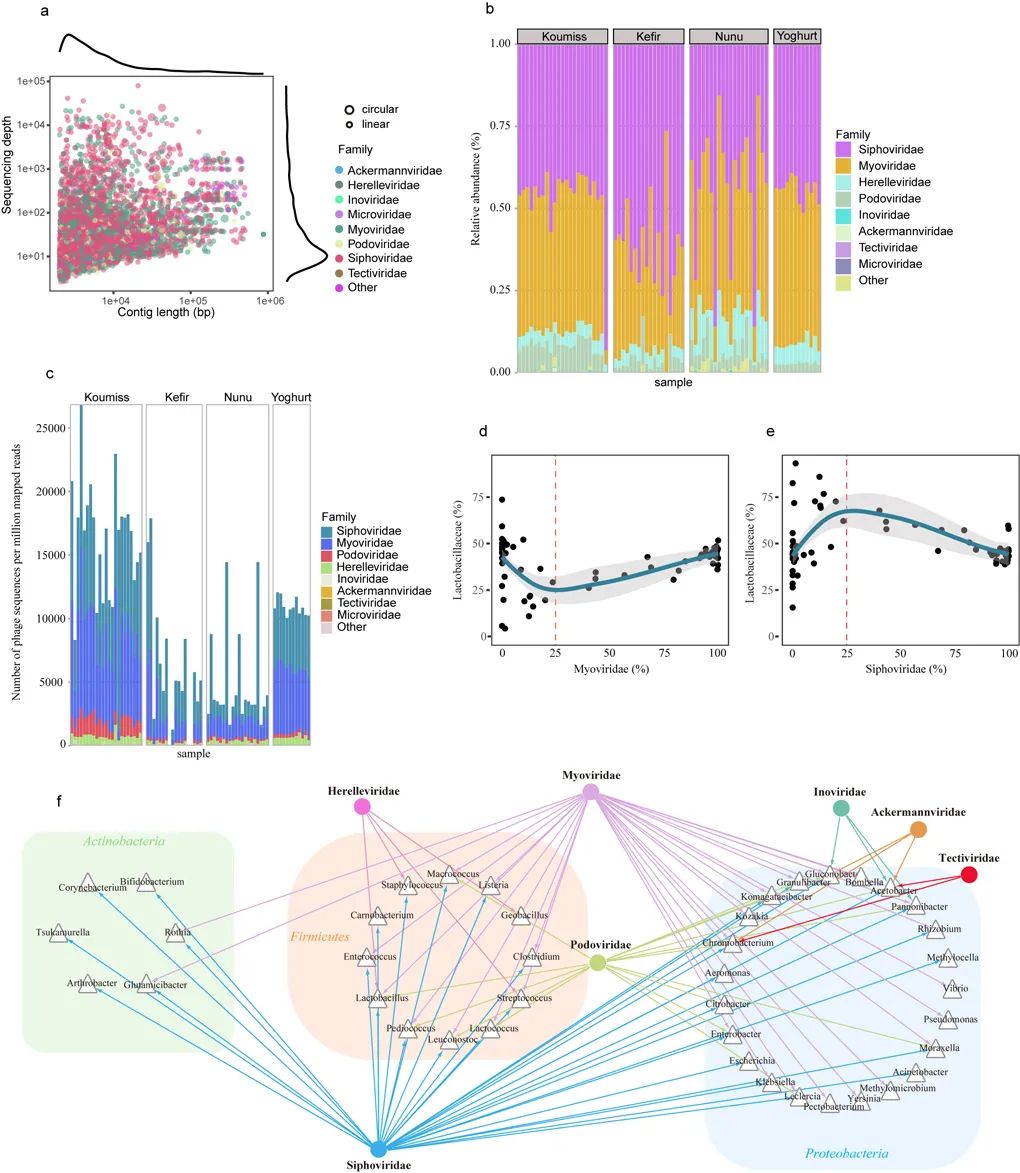
传统发酵乳制品菌群中的噬菌体
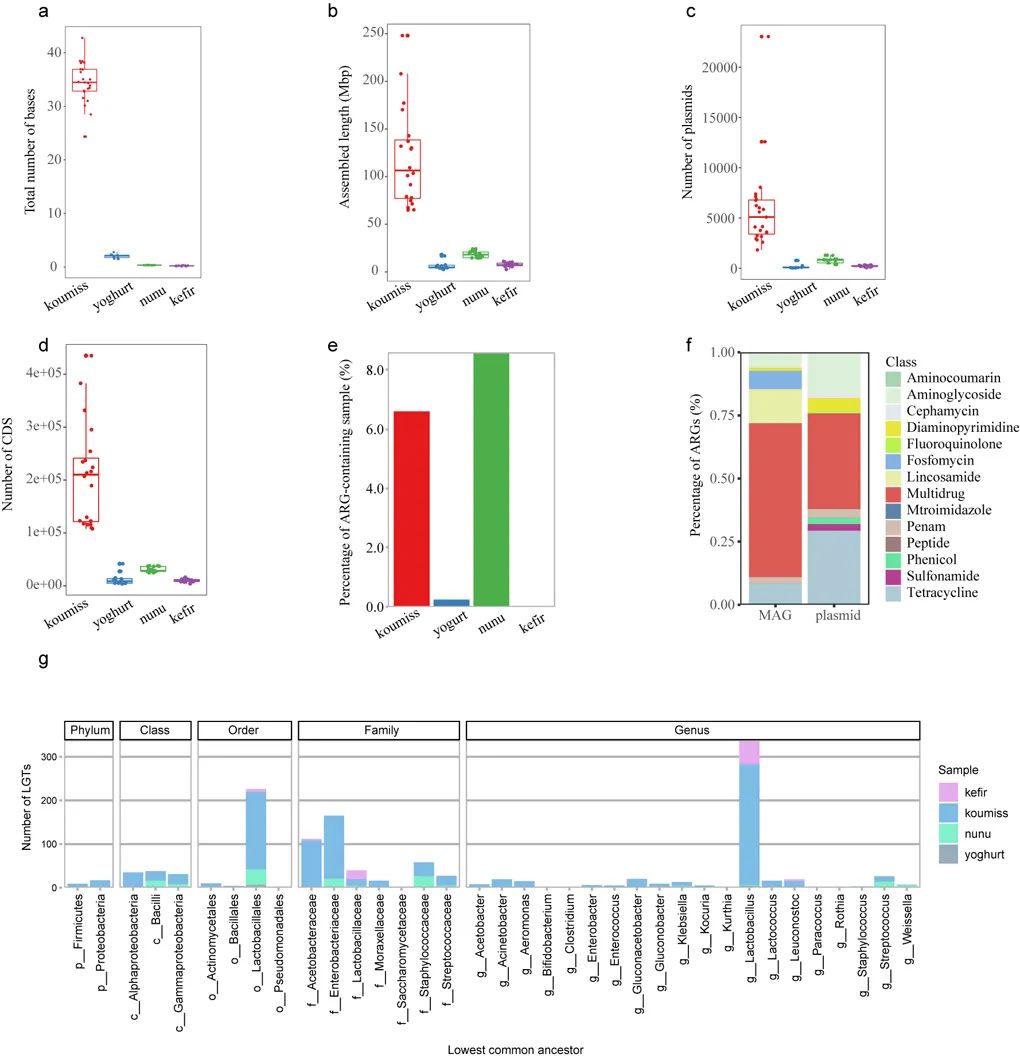
传统发酵乳制品的宏基因组特征
免责声明:本文仅代表作者个人观点,与中国益生菌网无关。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。
版权声明
1.本站部分转载的文章非原创,其版权和文责属于原作者。2.本网所有转载文章、链接及图片系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与我们联系,对可以提供充分证据的侵权信息,bio149将在确认后12小时内删除。3.欢迎用户投递原创文章至86371366@qq.com,经审核后发布到首页,其版权和文责属于投递者。







