利用单细胞扩增技术对酸马奶微生物组的研究
时间:2017-10-20 来源:益生之源 作者:姚国强 惠文彦 浏览次数:2457摘要:酸马奶具有悠久的制作和食用历史,是乳酸菌等微生物资源的宝贵载体。本研究基于单细胞扩增技术对酸马奶进行宏基因组分析。通过分析30份酸马奶单细胞悬液,共鉴定出24种细菌。除酸马奶中常见的优势细菌外,如Lactobacillus helveticus、Lactococcus lactis、Lactobacillus buchneri、Lactobacillus kefiranfaciens 和Acetobacter pasteurianus,还成功鉴定出3个低丰度物种(Lactobacillus otakiensis、Streptococcus macedonicus和Ruminococcus torques)。功能基因组分析表明,酸马奶中细菌编码丰富的氨基酸转运和代谢及碳水化合物转运和代谢基因,特别是含有大量的乳糖代谢基因,这些基因对酸马奶中细菌的生长繁殖及其特有风味的形成具有重要意义。该研究为传统发酵乳中微生物多样性及功能基因解析提供了新的思路和方法。
关键词:酸马奶;单细胞扩增;宏基因组学;细菌多样性
1 引言
传统发酵乳制作和食用历史悠久且种类繁多,是分离、保藏乳酸菌等微生物资源的宝贵载体,其中酸马奶是研究微生物多样性的典型代表。过去几十年间,研究人员采用不同的纯培养方法对传统发酵乳微生物多样性进行了研究,然而,传统培养技术不能准确反映微生物之间的系统发育关系。
宏基因组学避免了传统方法在多样性研究中的局限性,可以更准确地反映发酵乳中微生物组成,已被广泛用于传统发酵乳中微生物多样性的研究。但基于“焦磷酸测序技术”的宏基因组学研究依然存在不足,只能重构生态环境中具有生长优势的微生物信息,无法克服这种方法学在细菌多样性研究过程中所面临“量”的瓶颈问题。单细胞扩增技术的应运为生物多样性的研究带来了新的手段,可用于获取难以培养微生物的遗传信息,在多样性研究方面蕴藏着巨大潜力。
本研究自传统发酵乳酸马奶中获取细菌单细胞(100个细胞/份),通过MDA扩增法进行单细胞全基因组扩增。采用二代高通量测序技术(Illumina)分析细菌的全基因组信息,解析酸马奶中细菌多样性及其编码的功能基因。
2 材料与方法
2.1 主要试剂与仪器
QIAGEN REPLI-g Single-Cell Kit 试剂盒,德国Qiagen公司;TruSeq DNA 小片段建库试剂盒,美国Illumina公司;ECLIPSE TS100倒置显微镜,日本Nikon公司;CFX 96 荧光定量PCR 仪,美国Bio-Rad公司;Illumina HiSeq 2500 测序平台,美国Illumina 公司。
2.2 样品采集
本研究共采集了10份酸马奶样品,其中5份采集自蒙古国地区,5份采集自中国内蒙古地区。将10份酸马奶样品通过单细胞处理后获得30份酸马奶单细胞悬液,进行后续的测序分析。
2.3 单细胞全基因组扩增(MDA法)
收集到的酸马奶样品通过连续梯度稀释获得酸马奶单细胞悬液(100个细菌/份), 使用QIAGEN REPLI-g Single Cell Kit试剂盒完成全基因组扩增。
2.4 文库构建及测序
取扩增产物DNA 作为模板,根据TruSeq DNA Sample Preparation Guide(Illumina,15026486 Rev.C)方法及流程进行文库制备文库构建,确保文库的有效摩尔浓度大于10 nM。检测合格的文库,在HiSeq 2500测序平台上运行双端测序程序(PE125),得到125 bp读长的双端序列reads,进行后续数据分析。
2.5 数据处理
2.5.1 数据过滤与比对
原始下机数据(Raw Data)进行数据过滤,去除接头污染的Reads和低质量的Reads;去除含N比例大于5%的Reads后对其进行质量、Q30统计、数据量统计和碱基含量统计等。利用基因组比对软件BWA进行比对去除宿主序列污染。
2.5.2 物种及多样性分析
采用Metaphlan软件进行物种含量统计和制图。为了更好的展现每个样本中物种的组成,采用GraPhlAn软件绘制系统发育树。分析Alpha多样性和Beta多样性为了比较样本内部和样本间物种的多样性。
2.5.3 基因组装、预测及注释
采用Velvet软件并结合自身开发的软件分析。从Scaffold组装序列长度、N50/N90序列长度、组装序列GC含量等方面全面统计本次序列组装情况。采用MetaGeneMark软件进行分析。使用Cd-Hit软件比较每个样本中基因的表达情况,并去除冗余基因,进行基因相对丰度分析,以便后续基因差异分析。
基因功能注释:为了分析预测出来的基因功能,使用BLAST软件将基因序列注释到功能数据库中。
3 结果与分析
3.1 测序质量评估
酸马奶单细胞悬液通过MDA扩增后,每个酸马奶样品获得大于5 Gb的测序信息。30份酸马奶单细胞悬液样品共计获得1,040,323,864个原始序列,平均每个样品含有 34,677,462个序列。经过滤去除不合格的序列后共,每100个细胞获得1,018,381,702个有效序列。从图1可以看出,在此测序水平下,样品中细菌的多样性已能充分展现。

3.2 分类注释
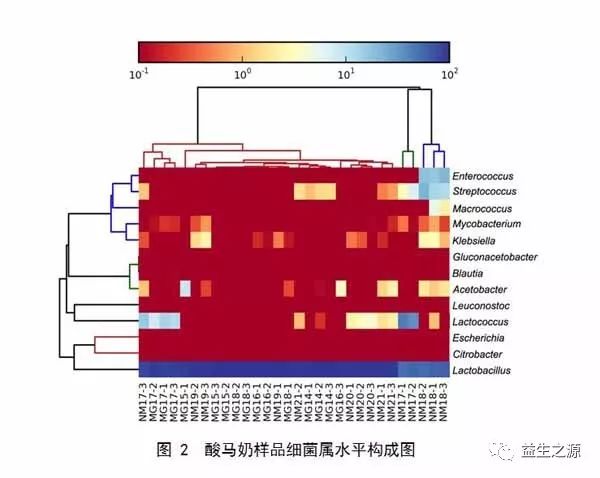
参考已发表的有关酸马奶生物多样性的报道,我们将以前报告的属和种归为常见属和种,而未曾报告的微生物类群归为低丰度物种。本次研究的酸马奶中共鉴定出13个细菌属(图2),乳杆菌属(Lactobacillus)是所有酸马奶中的优势菌属,含量介于52.721%~99.983%,平均相对含量为89.632%;其次为乳球菌属(Lactococcus)(平均相对含量为4.791%)和链球菌属(Streptococcus)(平均相对含量为2.140%)。
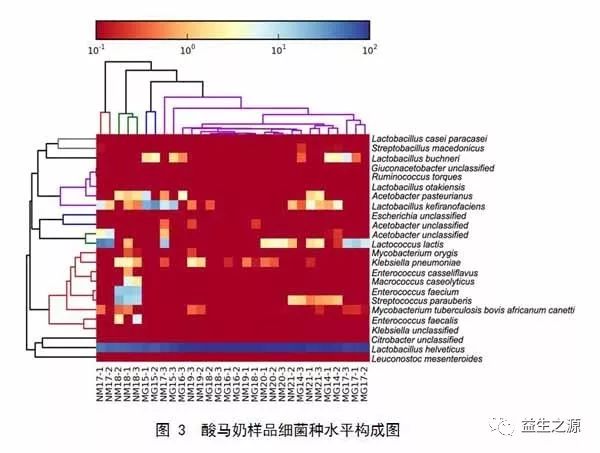
酸马奶中共鉴定出24种细菌(图3),蒙古国和内蒙古地区的酸马奶样品中共同种为Lactobacillus helveticus,Lactococcus lactis,Lactobacillus kefiranofaciens,Streptococcus parauberis和Enterococcus faecium,且其平均含量均>1.000%。Lactobacillus helveticus是来自蒙古国和内蒙古地区所有酸马奶中的优势种,平均含量为(85.530±6.380)%。其他种相对含量低于1.000%,极个别含量低于0.001%,其中Lactobacillus otakiensi、Streptococcus macedonicus和Ruminococcus torques是低丰度物种。
酸马奶中除检测到乳酸菌和醋酸菌等常见细菌外,还检测到微量的病原菌(如:Klebsiella pneumonia和Mycobacterium orygis)以及其他具有生物学应用潜力的物种(如:Macrococcus caseolyticus)。这些研究结果不仅有利于全面了解酸马奶中的细菌组成,而且为发酵乳生产过程中的细菌污染检测提供了新的方法。
3.3 功能基因注释
30份酸马奶单细胞悬液宏基因组信息COG注释如图4所示。通过酸马奶细菌COG基因注释,我们发现酸马奶中细菌含有丰富的氨基酸转运和代谢(E)及碳水化合物转运和代谢(G)基因。
乳糖是马奶中的主要碳水化合物,也是细菌的重要代谢能量来源,为了解其代谢机理,解读了与乳糖代谢相关的基因信息(表1)。在酸马奶发酵过程中,乳糖和部分半乳糖通过转运系统(lacFE)吸收进入细菌细胞内,并伴随磷酸化被水解成葡萄糖和半乳糖-6磷酸;抑或经β-半乳糖苷酶(lacZ)切断半乳糖苷键,水解成葡萄糖和半乳糖。葡萄糖通过糖酵解途径(EMP)代谢为乳酸,而在大多数菌株中,半乳糖不能被直接代谢,需通过反转运系统排出到外部环境中,重新进入其他微生物细胞内分解成半乳糖6-磷酸。半乳糖6-磷酸通过塔格糖途径(lacABCD)代谢成磷酸丙糖(甘油醛-3-磷酸和二羟丙酮磷酸),该途径由半乳糖6-磷酸异构酶 (LacAB),D-塔格糖6-磷酸激酶(LacC)和塔格糖1,6-二磷酸醛缩酶(LacD)组成。

表 1 酸马奶样品宏基因组分析中有关乳糖代谢的相关基因

同其他类型的发酵乳一样,酸马奶风味的产生依赖于氨基酸转化途径,从本研究的数据中筛选出编码游离氨基酸分解代谢相关途径的基因。发现潜在的针对精氨酸,天冬氨酸,甲硫氨酸和异亮氨酸特异性氨基转移酶类的种类,经鉴定来自低丰度Streptococcus macedonicus种的class I/class II(IPR004839)。进一步比对分析氨基酸裂合酶类,发现许多信息,包括S-核糖基高半胱氨酸裂解酶(S-ribosylhomocysteine lyase),精氨基琥珀酸裂解酶(argininosuccinate lyase),天冬氨酸氨裂解酶(aspartate ammonia-lyase),胱硫醚γ-裂合酶(cystathionine gamma-lyase),组氨酸氨裂解酶(histidine ammonia-lyase)和O-乙酰高丝氨酸(硫醇)裂解酶(O-acetylhomoserine (thiol)-lyase)。
4 结论
(1)基于单细胞扩增技术的Illumina测序分析发现,酸马奶样品中细菌归为7个门、13个属和24个种,其中优势属为Lactobacillus、Lactococcus和Streptococcus。Lactobacillus helveticus、Lactococcus lactis和Lactobacillus kefiranofaciens为酸马奶优势种,Lactobacillus otakiensi、Streptococcus macedonicus 和Ruminococcus torques是低丰度物种。
(2)酸马奶中细菌编码丰富的氨基酸转运和代谢及碳水化合物转运和代谢基因,特别是含有大量的乳糖代谢基因,这些基因对酸马奶中细菌的生长代谢及其特有风味的形成具有重要意义。
5 展望
采用基于单细胞扩增技术的基因组学方法分析了来自蒙古国和内蒙古地区酸马奶样品中的细菌多样性及其功能性基因组,特别是检测到多种低丰度物种。研究首次证明了将单细胞扩增技术纳入细菌生物多样及污染微生物检测的可行性。该研究可进一步优化当前的方法,如增加DNA扩增前的样品稀释度,以增加发现罕见和新型细菌的机率。此外,也可以使用单分子实时测序技术来改进基因组组装过程。
文献来源:Guoqiang Yao, Jie Yu, Qiangchuan Hou, Wenyan Hui, wenjun Liu, Laiyu Kwok, Bilige Menghe, Tiansong Sun, Heping Zhang, Wenyi Zhang. A perspective study of bacterial diversity in koumiss by metagenomics analysis based on single-cell amplification technique[J]. Frontiers in Microbiology,2017,8:165.
免责声明:本文仅代表作者个人观点,与中国益生菌网无关。其原创性以及文中陈述文字和内容未经本站证实,对本文以及其中全部或者部分内容、文字的真实性、完整性、及时性本站不作任何保证或承诺,请读者仅作参考,并请自行核实相关内容。
版权声明
1.本站部分转载的文章非原创,其版权和文责属于原作者。2.本网所有转载文章、链接及图片系出于传递更多信息之目的,且明确注明来源和作者,不希望被转载的媒体或个人可与我们联系,对可以提供充分证据的侵权信息,bio149将在确认后12小时内删除。3.欢迎用户投递原创文章至86371366@qq.com,经审核后发布到首页,其版权和文责属于投递者。







